土壤健康是维护生态系统功能和农业生产力的基础,监测和评估土壤健康对确保生态和食品安全至关重要。目前大多数土壤健康研究主要集中在重金属和农药残留等污染物的检测和分析,忽视了生物群落要素。为验证eDNA方法能否有效识别不同农业生态系统的群落变化和健康状况,我院张远教授团队利用eDNA技术在浙江省嘉善县开展了典型农业生态系统土壤生物监测研究,相关成果近期发表在Environmental Research期刊上。该论文第一作者为硕士研究生幸凯航,通讯作者为李飞龙副教授。

本研究采用eDNA技术对嘉善县4个典型农业系统(即经济作物田、鱼塘、藕塘和水稻田)土壤生物区系进行监测(图1),筛选出48个敏感生物指标,建立了以生物多样性指标为核心的土壤生态健康评价框架。然后,与传统的基于理化变量的土壤质量指标(SQI)进行比较,评价新框架在农业生态系统中的适用性和可靠性。主要研究目的包括:(1)研究不同农业生态系统土壤生物的种类组成,分析生物多样性及其环境胁迫的决定因素;(2)筛选敏感生物指标,建立土壤生态健康评价新框架;(3)比较新型eDNA指数与传统理化SQI指数,确定eDNA在土壤生态健康评价中的适用性。
研究结果
利用eDNA技术,在不同农业生态系统的土壤样品中监测到多个类群的生物组成,包括真菌、细菌、后生动物以及其他真核微生物(图2)。eDNA技术揭示了土壤生物多样性的新视角,使能够以前所未有的精度和深度监测土壤生物多样性。

图2.监测的属或种在不同农业生态系统和季节的相对丰度存在显著差异。气泡大小表示该物种在所有土壤样品中监测到的属或种的百分比。
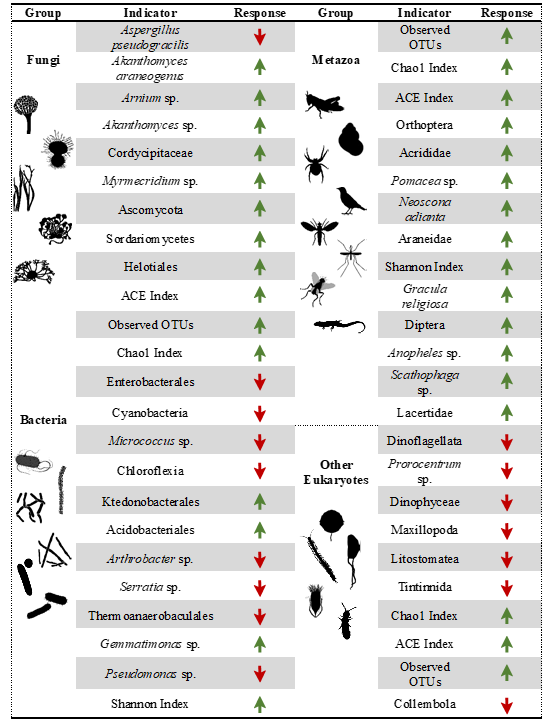
对土壤理化变量进行主成分分析(PCA),构建代表新的理化变量描述符的环境应力梯度。通过Spearman相关系数矩阵检验多样性指数与土壤理化变量之间的关系,筛选出了48个能够响应环境胁迫变化的敏感生物指标(表1)

表1.与土壤理化变量显著相关的生物指标。绿色箭头表示正相关,红色箭头表示负相关。
研究发现,通过四个类群的eDNA指数与SQI指数拟合后,四种类群eDNA指数均与SQI指数有显著的相关性(P<0.01),其中细菌和后生动物eDNA指数与SQI指数的相关性最明显(R2bacteria = 0.50, R2 metazoa = 0.58,图3),表明构建的以生物指标为核心的土壤生态健康指数具有评价土壤环境污染的能力。
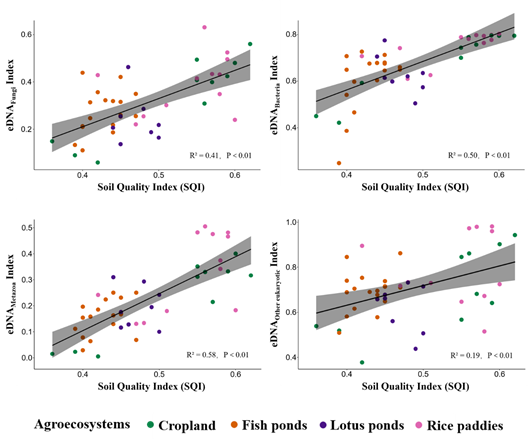
图3.四个类群的eDNA指数与传统土壤质量指数(SQI)的比较。黑色实线来自多元线性回归,阴影区域表示95%置信区间。
研究基于eDNA技术监测了嘉善典型农业生态系统的土壤生物多样性,联合理化性质筛选敏感性生物指标,构建eDNA指数评价体系并与SQI指数对比后发现:eDNA指数与SQI指数具有显著的相关性,这一发现说明基于生物指标的eDNA指数具有评价土壤生态健康的能力,凸显了eDNA技术在未来土壤健康研究领域中的潜力。
Xing K, Lu W, Huang Q, et al. Soil eDNA biomonitoring reveals changes in multitrophic biodiversity and ecological health of agroecosystems[J]. Environmental Research, 2024: 119931.
https://doi.org/10.1016/j.envres.2024.119931

 粤ICP备05008833号
粤ICP备05008833号